ggplot2でなんでもプロット(table編) #RAdventJP
R Advent Calendar 2013の2日目です。
今回のネタは、本当は去年どっかでやろうと思ってたものだったんですが、機を逃して温めすぎて発酵しかけてたものです(このネタの存在自体忘れてた)。とりあえず、日本語での情報は少なくともまだウェブには無いようなので安心。ちなみにtable編と書いていますが、続編の予定はありません。もちろん、興味を持っていただいた方は好きに引き継いでもらってかまいません。
fortifyとは?
さて、ggplot2を利用されている方は多いと思いますが、高機能な分みなさん使いこなすのに苦労されているようです。実際一朝一夕で使えるようにはならないものではあるんですが、実際には便利な機能があるのにそれを知らないために苦労してただけ、ということもあったりします。今回紹介するのは、そんな便利機能の中でも特に知名度が低いと思われるfortify()という機能(関数)です。
"fortify"という単語自体が聞き慣れないですが、「砦」を意味する"fort"からの派生語です。「要塞化する」といったような物々しい意味なのですが、もう少し抽象的に、「強化する」というぐらいの意味でも用いられるようです。
では、fortify()は何で何を強化するのでしょうか。答えはfortify()のヘルプのタイトルにあります。
Fortify a model with data.
「モデルでデータを強化する」ということですね。重回帰分析を例に出せば、重回帰分析によって得られた予測値や残差といったような情報を、元のデータフレームに付け足す、というのがfortify()の機能です。ここで一つ注意すべき点として、fortify()の仕様的には何らかのオブジェクトを受け取りデータフレームを返す、ということしか必要とされていない、ということです。つまり、「モデルでデータを強化する」という名目に反してデータは実は必須ではなく、「モデル(オブジェクト)をデータフレームに変換する」というのがfortify()の本質です。
fortifyの使い途
fortify()自体はsummary()等と同じく総称的関数となっており、実際に変換するためにはクラスごとにメソッドを用意する必要があります。じゃあ、なんでわざわざそんな総称的関数が必要なんでしょうか。例に挙げた重回帰分析の予測値や残差のプロットなんて、fortify()を知らなくても多くの人がやっていることです。にもかかわらずそんな一見面倒なものが用意されているのは、ggolot2でデータフレーム以外のオブジェクトも柔軟にプロットできるようにするためです。ggplot()のdata引数に渡されたオブジェクトには必ずfortify()が適用されます。そして、fortify()を通せば必ずデータフレームが返ってくることになっています。つまり、ggplot()は受け取ったデータをとりあえずfortify()に丸投げして、可能であればデータフレームに変換して返してもらう、ということをしているわけです。
ggplot2はデータフレームしかプロットに使えないのが不便、と思っている人は多いのではないでしょうか。実際には、上記の仕組みによって、クラス専用のfortifyメソッドを用意してやれば、ggplot2はなんでもプロットできるようになっています*1。
tableをggplot2で使う
さて、ようやく本題です。タイトルの通り、tableをggplot2で使えるようにしてみましょう。まずは適当なデータを突っ込んでみます。
> library(ggplot2) > t1 <- as.table(Titanic[1:3,,2,2]) > p1 <- ggplot(data=t1) Error: ggplot2 doesn't know how to deal with data of class table
怒られました。「ggplot2はtableクラスのデータをどう処理すればいいのか知りません」といっています。そこで、table用のfortifyメソッドを定義してみましょう。
fortify.table <- function(model, ...) { data <- reshape2::melt(model) return(data) }
実質一行ですね。これはreshape2パッケージのmelt()を使って*2以下のような変換を行っています。reshape2が必要ですが、reshape2はggplot2の依存パッケージなので、ggplot2がインストールされていれば必ずインストールされているはずです。
> t1
Sex
Class Male Female
1st 57 140
2nd 14 80
3rd 75 76
> reshape2::melt(t1)
Class Sex value
1 1st Male 57
2 2nd Male 14
3 3rd Male 75
4 1st Female 140
5 2nd Female 80
6 3rd Female 76さて、再チャレンジです。
> p1 <- ggplot(data=t1)
次は怒られませんでした。では、ちょっと付け足してプロットしてみましょう。
> p1 + geom_bar(aes(Sex, value, fill=Class), > stat="identity")
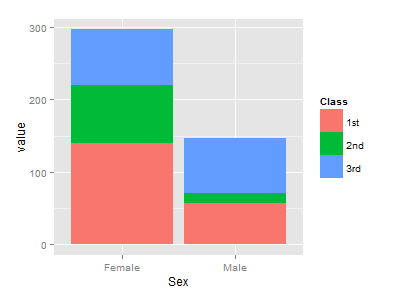
何の変哲も無い棒グラフがプロットされました。
> p1 + geom_bar(aes(Sex, value, fill=Class), > stat="identity", > position="fill") + coord_flip()
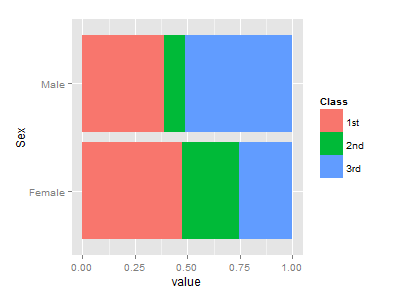
ちょっと付け足して帯グラフにしてみました。
まとめ
実質的には変換用関数で変換する一手間が減るだけなのですが、案外便利です。もっとも、fortify()の意義は、クラス作成者自らがfortifyメソッドを定義しておくことで、ユーザーが変換の手間をかけずにオブジェクトをggplot2に使えるようになる、というところにあるのではないかと思います。ユーザーがggplot2を使いそうなパッケージを作っている方は、fortifyメソッドを合わせて用意することを考えてみてはいかがでしょうか。
おまけ
ggplot2にデフォルトで用意されているfortifyメソッド一覧と一例としてfortify.lm()の中身、および今回のスクリプトをまとめたものです。
> methods(fortify)
[1] fortify.cld* fortify.confint.glht*
[3] fortify.data.frame* fortify.default*
[5] fortify.glht* fortify.Line*
[7] fortify.Lines* fortify.lm*
[9] fortify.map* fortify.NULL*
[11] fortify.Polygon* fortify.Polygons*
[13] fortify.SpatialLinesDataFrame* fortify.SpatialPolygons*
[15] fortify.SpatialPolygonsDataFrame* fortify.summary.glht*
Non-visible functions are asterisked
> getAnywhere("fortify.lm")
A single object matching ‘fortify.lm’ was found
It was found in the following places
registered S3 method for fortify from namespace ggplot2
namespace:ggplot2
with value
function (model, data = model$model, ...)
{
infl <- influence(model, do.coef = FALSE)
data$.hat <- infl$hat
data$.sigma <- infl$sigma
data$.cooksd <- cooks.distance(model, infl)
data$.fitted <- predict(model)
data$.resid <- resid(model)
data$.stdresid <- rstandard(model, infl)
data
}
<environment: namespace:ggplot2>